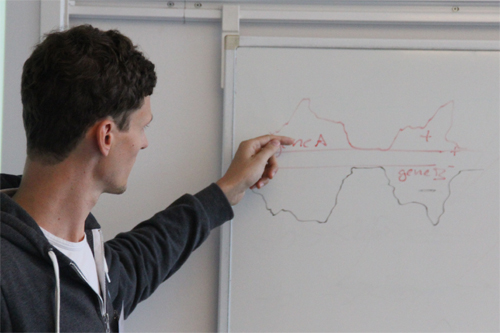
NGS Inhouse-Schulung und Vor-Ort-Schulungen
Firmeninterne Schulungen, individuelle Schulungen und Schulungen vor Ort mit speziellen Kursinhalten für Ihre Anforderungen
Neue Sequenziertechnologien ermöglichen ein extrem schnelles Auslesen genomischer Informationen (DNA) in immensem Umfang bei gleichzeitig hoher Qualität. Über 50 konkrete biologisch-medizinische Fragestellungen lassen sich mit Hilfe von Next-Generation Sequencing (NGS) beantworten. Entsprechend verbreiten sich die Maschinen rasant im Bereich der Forschung, der Diagnostik und zunehmend auch im klinischen Umfeld. Komplexe bioinformatische Verfahren sind notwendig, um die dabei anfallenden Daten auszuwerten und in nutzbare Erkenntnisse zu überführen.
ecSeq Bioinformatics GmbH bietet seit 2012 öffentliche Kurse und Inhouse-Schulungen im Bereich der NGS Bioinformatik an. Ihr Team wählt bei einer Inhouse-Schulung die für Sie relevantesten Themen aus. Zusammen mit Ihnen entwickeln wir das perfekte Curriculum und passende praktische Übungen, sodass Sie nicht nur ein tiefes Verständnis der Sequenziertechnologien und Analysemöglichkeiten gewinnen, sondern auch in die Lage versetzt werden, Sequenzierdaten selbständing auszuwerten. Mit über 50 Publikationen und andauernden Forschungsprojekten im Bereich NGS stellen wir sicher, dass unsere Trainer aktuelle und relevante Informationen liefern können. Nutzen Sie die Erfahrung aus dutzenden durchgeführten NGS Schulungen und aktuelles Wissen aus Bioinformatik Forschung für Ihre Organisation.
Unsere Philosophie
Training von Experten
Alle unsere Trainer sind Experten in ihrem jeweiligen Gebiet. Durch langjährige Mitarbeit an Forschungs- oder Industrieprojekten haben sie eine unvergleichliche Expertise erlangt. Im schnelllebigen Gebiet der NGS Datenanalyse ist aktuelles Wissen unabdingbar.
Open-source NGS Programme
Wir nutzen nur open-source Programme, die jeder frei verwenden kann. In unseren Kursen versuchen wir verschiedene Programme (z.B. NGS-Mapper) zu verwenden und zu vergleichen. Je nach Fragestellung sind unterschiedliche Programme empfehlenswert.
Effektives Lernen mit individuellem Kursmaterial
Um ein optimales Lernumfeld zu erzeugen, haben wir unsere Kursmaterialien und Beispieldaten sorgfältig entwickelt und an die Bedürfnisse von Anfängern angepasst. Der praktische Teil des Kurses wird auf einem Linux-System durchgeführt, das sich auf einem bootbaren USB-Stick befindet. Nach dem Kurs bekommt jeder Teilnehmer seinen Stick und kann damit weiterarbeiten.
Vorteile unserer Inhouse-Schulungen
- Sie entscheiden, welche Kursinhalte Sie wünschen
- Sie bestimmen die Größe der Gruppe
- Sie können Ihr eigenes Computersystem oder Ihre eigenen Daten verwenden
- Sie bekommen professionelle Hilfe direkt vor Ort
- Sie sparen Geld durch wegfallende Reisekosten
- Sie gewinnen Zeit (kein Anreise- und Abreisetag)
Kursinhalte
Vielfältige Anwendungsgebiete
- Genetische Tests
- Diagnostik von Erbkrankheiten
- Pharmakogenomik
- Pränatales und Neugeborenen-Screening
- Forensik
- Detektion von Biomarkern und differentiell exprimierter Gene
- Untersuchungen der Biodiversität und Metagenomik
- Genom-Assemblierung
- Epigenetik
Sequenziertechnologien
Methodik, Vorteile und Fehlerquellen werden vorgestellt:- (alt) Sanger Sequenzierung
- Illumina Sequenzierung
- 454 Sequenzierung
- Ion Torrent Sequenzierung
- PacBio Sequenzierung
- Nanopore Sequenzierung
Verschiedene Protokolle
- DNA-Seq
- RNA-Seq
- small RNA-Seq
- Exome-Seq
- ChIP-Seq
- Bisulfite-Seq
Grundlagen der NGS Datenanalyse
- Datenformate für Sequenzen (FASTQ)
- Durchführen von Qualitätskontrolle und Vorverarbeitung (Adapter Clipping, Trimming)
- Einführung in Read Alignment
- Vergleich Alignment Tools wie BWA, Bowtie2, TopHat
- Datenformate für Alignments (SAM- / BAM-Format)
- Die Nutzung wichtiger NGS-Werkzeuge wie samtools, BEDtools
- Auswertung der Alignments, Mapping Statistiken
- Visualisierung der Alignments
Differentielle Expression Analyse mit RNA-Seq
- RNA-Seq Alignment (STAR, TopHat)
- Quantifizierung der Expression von Genen, Isoformen und Exonen
- Statistik der differentiellen Expression (DE)
- Praktische DE-Analysen mit Bioconductor
Technische Herausforderungen
- Welche Infrastruktur / wieviel Speicher für NGS-Analysen benötigt wird
- Welche Software bzw. welche Werkzeuge sich für NGS-Anwendungen verwenden lassen
- Wie sich NGS Auswertungen automatisieren lassen
Interesse geweckt?
Falls Sie Interesse an einer Inhouse-Schulung haben, verwenden Sie bitte den folgenden Button um eine Anfrage zu stellen, oder schreiben Sie eine E-Mail an  . Einer unserer Mitarbeiter wird sich dann schnellstmöglich mit Ihnen in Verbindung setzen und zusammen mit Ihnen ein individuelles Kursprogramm zusammenstellen.
. Einer unserer Mitarbeiter wird sich dann schnellstmöglich mit Ihnen in Verbindung setzen und zusammen mit Ihnen ein individuelles Kursprogramm zusammenstellen.
Kundenmeinungen
"As someone who was previously terrified of the command line, this was a terrific course to build confidence in navigating the terminal. Moreover, I walked away with a much deeper understanding of sequencing chemistry and the relative merits of different software packages for analysing different types of sequencing data. I highly recommend this course for any wet lab biologist whether at the student, postdoc, or PI level!" Alexandra McCorkindale, Max Delbrueck Center for Molecular Medicine
"Great Training! Competent trainers and enjoyed the practical part of the training very much." Joerg Schmiedle, F. Hoffmann-La Roche AG
"As a wet lab biologist, I have been dabbling in analysing my RNA-seq data myself. This course gave me a lot of context about mapping and differential expression software that I hadn't been able to grasp on my own. This knowledge will certainly improve the quality of my analyses. Additionally, I learned a lot of handy Linux commands and tricks that make my life a lot easier." Paul Essers, Max-Planck-Institute for the Biology of Ageing
"Good resume of the latest technologies and applications. Definitely will improve the communication with our bioinformatics colleagues for performing the proper analysis and understanding of the data. Thanks for an intensive but nice course." Susana Gonzalez Fernandez-Nino, Bayer Crop Science